在蛋白质相互作用网络中挖掘蛋白质复合体的研究(附答辩)
来源:wenku7.com 资料编号:WK73192 资料等级:★★★★★ %E8%B5%84%E6%96%99%E7%BC%96%E5%8F%B7%EF%BC%9AWK73192
以下是资料介绍,如需要完整的请充值下载。
1.无需注册登录,支付后按照提示操作即可获取该资料.
2.资料以网页介绍的为准,下载后不会有水印.资料仅供学习参考之用. 密 保 惠 帮助
资料介绍
在蛋白质相互作用网络中挖掘蛋白质复合体的研究(附答辩)(含选题审批表,任务书,开题报告,中期检查表,毕业论文35000字,答辩记录)
摘 要:从大规模蛋白质相互作用网络中识别蛋白质复合体对预测蛋白质功能、解释特定的生物进程具有重要意义。本文首先分析了蛋白质相互作用网络的小世界性,在此基础上,分析并实现了几种经典蛋白质复合体挖掘算法,主要包括:基于密度的局部搜索算法(CPM算法和IPC-DM算法)和层次聚类算法(G-N算法、FAG-EC算法及其改进后的HC-Wpin算法)。最后对融合多元数据信息挖掘蛋白质复合体进行了简单的研究。
关键词:蛋白质相互作用网络;蛋白质复合体;小世界性;基于密度的局部搜索算法;层次聚类算法
The Research of Identifying Protein Complexes In Protein Interaction Networks
ABSTRACT:Identification of protein complexes from the large-scale protein interaction networks is significant for predicting protein function and explaining the specific biological processes. In the paper, we first analyze the "small-world" of protein-protein interaction networks. Then, we analyze and implement several classical algorithms for identifying protein complexes in protein interaction networks. These algorithms are CPM, IPC-DM, G-N, FAG-EC, and HC-Wpin. CPM and IPC-DM are density-based local search algorithms. G-N, FAG-EC, and HC-Wpin are hierarchical clustering algorithms. Finally, we introduce the research of identifying protein complexes based on multiple data.
KEY WORDS: protein interaction networks;protein complexes;small-world;density-based local search algorithm;hierarchical clustering algorithm
目 录
摘 要………………………………………………………………………………………1
关键词………………………………………………………………………………………1
1 前言………………………………………………………………………………………2
2 绪论………………………………………………………………………………………2
2.1 蛋白质相互作用网络…………………………………………………………3
2.2 蛋白质复合体与功能模块……………………………………………………5
2.3 本文的主要研究内容…………………………………………………………6
3 蛋白质相互作用网络的小世界性………………………………………………………7
3.1 小世界性的定义………………………………………………………………7
3.2 蛋白质相互作用网络的直径与特征路径长度的分析………………………8
3.3 蛋白质相互作用网络的聚集系数与模块化结构的分析…………………10
3.4 本章小结……………………………………………………………………10
4 基于密度的蛋白质复合体挖掘算法研究……………………………………………10
4.1 CPM算法及其改进算法…………………………………………………11
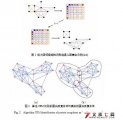
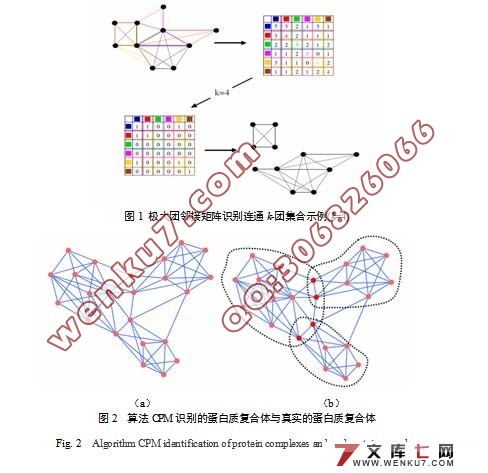
4.1.1 CPM算法…………………………………………………………12
4.1.2 CPM改进算法CPM-DR……………………………………………13
4.1.3 比较CPM算法和CPM-DR算法……………………………………14
4.2 DPClus算法及其改进算法……………………………………………………16
4.2.1 DPClus算法……………………………………………………17
4.2.2 IPC-DM算法………………………………………………………18
4.2.3 对DPClus算法和IPC-DM算法的结果分析………………………23
4.3 本章小结………………………………………………………………………25
5 基于层次聚类的蛋白质复合体挖掘算法研究………………………………………25
5.1 G-N算法……………………………………………………………………27
5.2 FAG-EC算法及其改进后的HC-Wpin算法……………………………………28
5.2.1 边聚集系数的引入……………………………………………………28
5.2.2 量化定义功能模块…………………………………………………29
5.2.3 算法FAG-EC…………………………………………………………30
5.2.4 加权层次聚类算法HC-Wpin………………………………………32
5.2.5 对FAG-EC算法和HC-Wpin算法的结果分析…………………34
5.3 本章小结………………………………………………………………………35
6 融合多元数据的蛋白质复合体挖掘研究……………………………………………35
6.1 融合基因表达数据的蛋白质复合体挖掘……………………………………36
6.2 融合其他多元信息的蛋白质复合体挖掘……………………………………37
6.3 蛋白质复合体挖掘的扩展研究………………………………………………38
6.4 本章小结………………………………………………………………………39
7 结束语…………………………………………………………………………………40
参考文献……………………………………………………………………………………42
致 谢………………………………………………………………………………………48
|